Synthèse d'images, ESSI3
Examen, 15 février 2004
Objectifs et moyens
L'objectif de ce travail sera de représenter des molécules sous la forme symbolique de leur formule chimique. Vous devrez définir les structures de données définissant les molécules et composer un graphe de scène pour les représenter. Des illustration du résultat attendu sont données ci-après.
Avant de commencer le travail de programmation, lisez le sujet dans son
entier et réfléchissez à la structure de votre programme.
Vous disposez d'installations linux sur CD avec Java3D, sa
documentation et les tutoriaux. Vous êtes
connectés sous l'identité 'knoppix'. Votre répertoire
utilisateur de travail (/home/knoppix) est un disque virtuel en
mémoire. Vous disposez d'une disquette pour la sauvegarde et le rendu
de votre travail. S'il est plus confortable de compiler et d'exécuter
le code depuis le répertoire de travail, cet espace mémoire ne survit
pas à un éventuel plantage machine. Il est donc indispensable de
copier régulièrement votre travail sur la disquette. Pour cela,
réalisez les commandes:
-
cp *.java /mnt/floppy pour ordonner la copie
-
sync pour forcer l'exécution immédiate de cette copie (les commandes d'accès disque étant en général retardées par le cache)
Java (JDK 1.5.0) et java3D (1.3.1) sont installés sur votre système. Vous devez configurer les variables d'environnement PATH et le CLASSPATH de votre système pour compiler et exécuter le code:
export PATH=/usr/java/jdk1.5.0/bin:${PATH}
export CLASSPATH=::/usr/java/jdk1.5.0/jre/lib/ext/j3dcore.jar:\
/usr/java/jdk1.5.0/jre/lib/ext/j3daudio.jar:\
/usr/java/jdk1.5.0/jre/lib/ext/j3dutils.jar:\
/usr/java/jdk1.5.0/jre/lib/ext/vecmath.jar
Ensuite, vous pouvez compiler en invoquant simplement la
commande javac et exécuter en invoquant
java. La documentation de java, java3D et les tutoriaux
sont installés localement sur votre système. Vous êtes cependant
isolés du réseau.
L'utilitaire emacs est installé sur vos machines. Vous
pouvez réaliser la compilation directement depuis emacs en utilisant
le menu 'Tools' -> 'Compilation'. Entrez la ligne de compilation
(javac *.java) lors de la première compilation. Vous
pouvez forcer la colorisation du code lors du premier chargement de
chaque fichier source avec la commande: 'Alt-x font-lock-mode'.
Un squelette de programme est disponible sur votre disquette dans le
fichier Example.java. Il ouvre un canvas 3D et affiche un
cube. Vous le modifierez et vous ajouterez les classes
nécessaires. Vous n'avez pas besoin d'étudier le détail du code
fournit. Il devrait vous suffire d'écrire vos classe et d'insérer les
appels à votre code dans le constructeur de la classe
Example.
Les 3 boutons apparaissant sous la fenêtre principale ont pour but de vous
faciliter la tâche si vous éprouvez le besoin d'utiliser des commandes
pour faciliter l'utilisation de votre programme. Afin de ne pas perdre
de temps en développement d'interface graphique, vous pouvez
simplement:
- Implémenter l'interface
ButtonListener dans la classe
qui doit recevoir les événements.
- Transmettre l'objet de réception des évènements au constructeur
de
Frame3D.
- Gérer les actions à associer à chaque bouton dans les méthodes
onButton1/2/3 de cette classe.
L'utilisation de ces boutons n'est en rien indispensable.
A la fin de la séance, vos fichiers seront récupérés depuis votre
disquette. Vous devrez y laisser:
- Un fichier README comprenant votre nom, la liste des classes
développées (avec pour chacune d'entre elles un bref descriptif de son
rôle) et la ligne de commande à exécuter pour lancer le programme. Vous
pouvez y ajouter un commentaire explicatif sur votre travail ou une
difficulté rencontrée qui vous a amené à proposer une solution particulière.
- La liste des fichiers sources (
*.java).
N'oubliez pas de copier tous les fichier du disque virtuel vers la
disquette avant de rendre votre travail!
Sujet
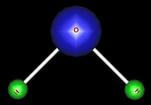
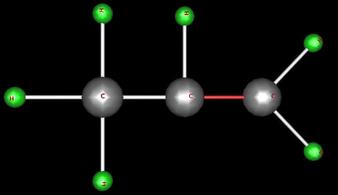
Vous représenterez des molécules chimiques simples constituées d'atomes d'hydrogène, de carbone et d'oxygène. Chaque atome est représenté par une sphère comme dans l'exemple suivant:

- Définissez une structure de données pour représenter les atomes. Chaque atome est décrit par un lettre, sa taille, sa couleur, et sa valence en respectant les données suivantes:
| Atome | Lettre | Taille relative | Couleur | Valence |
| Hydrogène | H | 1 | vert | 1 |
| Carbone | C | 2 | gris | 4 |
| Oxygène | O | 2.5 | bleu | 2 |
On rappelle que la valence d'un atome est le nombre (fixe pour un type d'atome particulier) de liaison que celui ci peut établir avec d'autres atomes. Des ensembles d'atomes liés entre eux forment ainsi des molécules. La valence est le nombre d'électron que des atomes liés partagent. Dans les exemples que vous manipulerez, chaque liaison peut être simple (partage d'un seul électron) ou double (partage de deux électrons).
- Donnez des représentations graphiques aux atomes sous forme de sphère de couleur. Affichez les trois types d'atome dans la fenêtre java3D.
- Donnez du recul à la caméra pour lui permettre de visualiser correctement la scène et ajoutez les lumières nécessaires.
- Ajoutez à l'affichage des sphères la lettre descriptive de chaque atome comme dans l'exemple ci-dessus. Avant de passer à la suite qui va nécessiter des modifications conséquentes, vous pouvez sauvegarder à part ce premier programme sous le nom 'Etape1.java'.
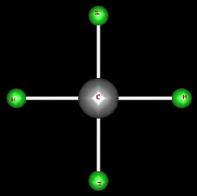
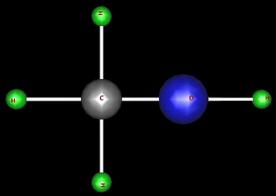
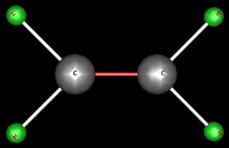
- Définissez une structure de données pour représenter une molécule. Les 6 molécules que vous allez représenter sont le dioxygène (O2), l'eau (H20), le méthane (CH4), le méthanol (CH3OH), l'éthylène (C2H4) et le propène (CH3CHCH2) qui sont représentées ci-après:
Par soucis de simplification, chacune de ces structures est décrite dans un plan et la distance entre 2 atomes liés est toujours la même. On peut donc représenter la position relative d'un atome et de son voisin par un simple angle (on prendra comme origine des angles l'axe X comme dans l'exemple suivant:
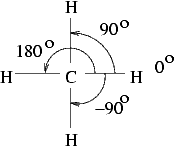 Mettez en oeuvre une structure aussi simple que possible pour la représentation des molécules. On pourra en particulier s'appuyer sur la structure arborescente des molécules. En tête de votre classe, insérez un commentaire expliquant la structure de donnée choisie.
Mettez en oeuvre une structure aussi simple que possible pour la représentation des molécules. On pourra en particulier s'appuyer sur la structure arborescente des molécules. En tête de votre classe, insérez un commentaire expliquant la structure de donnée choisie.
- Faîtes afficher les molécules comme des ensembles d'atomes répartis dans l'espace.
- Ajoutez les liaisons atomique à vos molécules, qui sont représentées comme des cylindres blanc joignant les atomes 2 à 2.
- Proposez un moyen pour différentier les liaisons doubles des liaisons simples. Trois liaisons doubles apparaissent dans les 6 molécules proposées: entre les deux atomes d'oxygène de l'O2, entre les deux atomes de carbone de l'éthylène, et entre deux des atomes de carbone du propène (les liaisons doubles peuvent facilement être devinées par la valence des molécules).
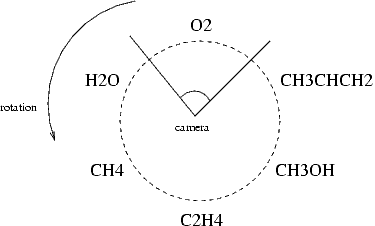
- Disposez les 6 molécules sur un cercle autour de la caméra et faites tourner celui-ci de manière à ce qu'elles défilent tour à tour devant la caméra:
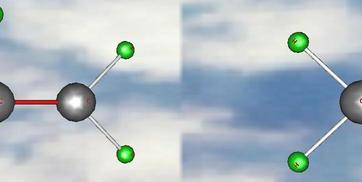
- Ajoutez une image de texture de fond à votre scène virtuelle (une image est disponible sur votre disquette):
- Déjà fini? Proposez une représentation compact des molécules (sans liaison, avec les atomes qui entrent en contact). Pouvez vous passer de manière progressive de la représentation compacte à la représentation liée et réciproquement?
- N'oubliez pas de copier tous les fichier du disque virtuel vers la
disquette avant de rendre votre travail!